Disminucion de dimensiones
The slides are available here!
Las técnicas de reducción de dimensión buscan simplificar la representación de los datos, sea eliminando atributos innecesarios (selección de características) o encontrando una proyección más compacta que mantenga la mayor parte de la información. A continuación, veremos métodos tanto supervisados (wrappers, filters) como no supervisados (PCA, entre otros).
Motivaciones para una Dimensión más Baja
Selección y Reducción de Atributos
Motivación General
- En muchos problemas de aprendizaje, hay atributos irrelevantes o redundantes.
- Pueden perjudicar el desempeño de un clasificador o incrementar el costo de entrenamiento.
- Dos enfoques principales:
- Selección de atributos (supervisado): escoger un subconjunto relevante para la tarea (clasificación, regresión…).
- Reducción de dimensionalidad (no supervisado): encontrar una proyección de menor dimensión que concentre la información de los datos.
Razones para Seleccionar Atributos
- Árboles de decisión:
- Aunque tratan de escoger atributos relevantes, si hay muchos atributos basura, puede aparecer sobreajuste (“aprender” ruido con árboles muy profundos).
- KNN:
- Se ve muy afectado por atributos irrelevantes, pues todas las dimensiones contribuyen igual al cálculo de distancias.
- Naïve Bayes:
- Robusto a atributos irrelevantes (los ignora en la probabilidad a posteriori), pero sufre con atributos muy correlacionados (redundantes).
En general, se quiere mitigar la curse of dimensionality (maldición de la dimensionalidad).
Selección de Atributos
Filtros vs. Envoltura
Feature-based (Filtros)
- Evalúan atributos por propiedades de los datos (p.ej. varianza, correlación, información mutua).
- Independientes del clasificador.
- Más rápidos y con menos riesgo de sobreajuste, pero no captan interacciones complejas con la tarea predictiva.
Model-based (Wrappers)
- Entrenan un clasificador sobre cada subconjunto para medir qué tan bueno es.
- Objetivo: maximizar la capacidad predictiva del modelo específico.
- Pueden ser computacionalmente costosos y propensos a sobreajuste, pero suelen encontrar subconjuntos de atributos más específicos a la tarea.
Métodos Feature-based (Univariate)
- Independientes de un modelo (univariate feature selection).
- Usan métricas generales (entropía, Information Gain, correlación, \(\chi^2\)…).
- Ejemplos:
- Information Gain (basado en entropía).
- Mutual Information.
- Correlation-based Feature Selection (CFS).
- Low variance.
Son rápidos y simples, pero no consideran cómo cada atributo interactúa con un clasificador concreto.
Mutual Information y \(\chi^2\) son útiles cuando los datos son dispersos (sparse).
Correlation-based Feature Selection (CFS)
- Se evalúa un subconjunto de atributos según correlaciones en los datos:
- Alta correlación con la clase.
- Baja correlación entre atributos (evitar redundancia).
- Se suele usar symmetric uncertainty para medir correlaciones entre atributos categóricos:
donde \(H(\cdot)\) es la entropía, e \(IG(A,B)\) la ganancia de información.
Búsqueda de subconjuntos
- El total de subconjuntos es \(\mathcal{O}(2^n)\).
- Se aplican heurísticas greedy (Forward selection, Backward elimination) o más avanzadas (Best-first search, Beam search, etc.).
Model-based (Scheme-Specific)
Selección basada en importancia de atributos
- Tras entrenar un modelo (ej. árbol, regresión lineal, random forest), se obtiene la importancia de cada atributo (coeficientes o feature importances).
- Se descartan los que no superan cierto umbral.
- SelectFromModel de scikit-learn permite automatizarlo.
Regularización \(\ell_1\) (Lasso)
- Induce parcimonia: algunos coeficientes se anulan.
- Ideal para selección en modelos lineales (regresión, logística).
- Penaliza \(\sum |w_j|\), forzando algunos \(w_j\) a 0.
- Integra fácilmente con
SelectFromModel.
Selección por Wrapper (Performance-based)
- Se prueban subconjuntos entrenando un modelo y midiendo su performance (accuracy, F1, AUC…).
- Muy costoso: reentrenar el modelo para cada subconjunto potencial.
- A menudo se hace greedy (Forward o Backward).
- Recursive Feature Elimination (RFE):
- Entrena estimador, extrae la importancia de atributos,
- Descarta los menos importantes,
- Repite con el subconjunto reducido hasta lograr la cantidad de atributos deseada.
Reducción de Dimensión
Más allá de “quitar” atributos, a veces transformamos los datos a una dimensión menor con un método no supervisado.
Intereses de la Reducción de Dimensión
- Simplificar los datos: Menos ruido, mejor generalización.
- Reducir costo computacional: Entrenar y predecir con menos dimensiones es más rápido.
- Visualización: Proyecciones 2D o 3D para entender la estructura de los datos.
- Evitar la curse of dimensionality: Métodos basados en distancia sufren en alta dimensión.
- Compresión: Reducir espacio de almacenamiento.
Análisis de Componentes Principales (PCA)
PCA es un método lineal que halla vectores propios de la matriz de covarianza. Permitten de representar la mayor parte de los datos, con menos vectores en la base del espacio.
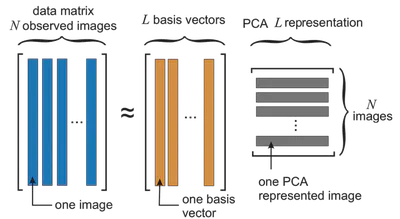

¿Por qué PCA?
- Reduce dimensión para análisis, visualización o compresión.
- Encuentra un sistema de coordenadas donde los ejes (componentes principales) corresponden a las direcciones de mayor varianza en los datos.
- Base calculada con las autovectores de la covarianza.
Ejemplo e Ilustración
- PCA ve la redundancia (varias variables correlacionadas) y la condensa en componentes que explican la mayor parte de la varianza.
- Datos \(\mathbf{X}_0\) centrados (o estandarizados) se proyectan en el subespacio donde la varianza proyectada es máxima.
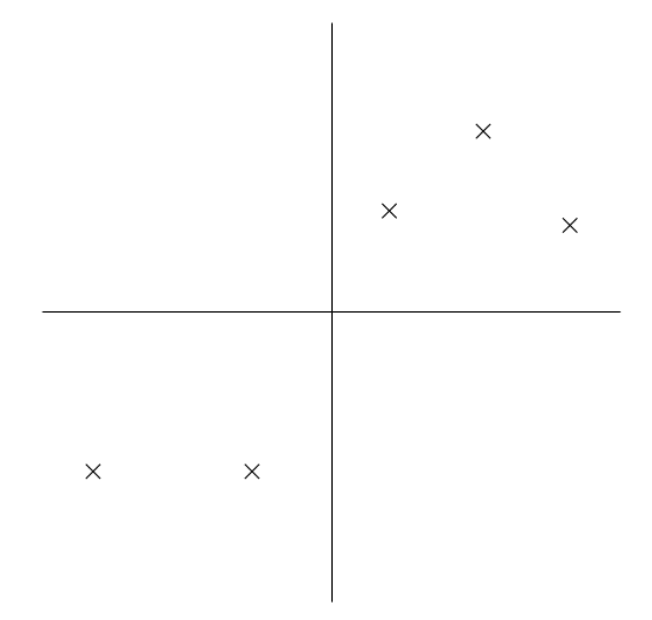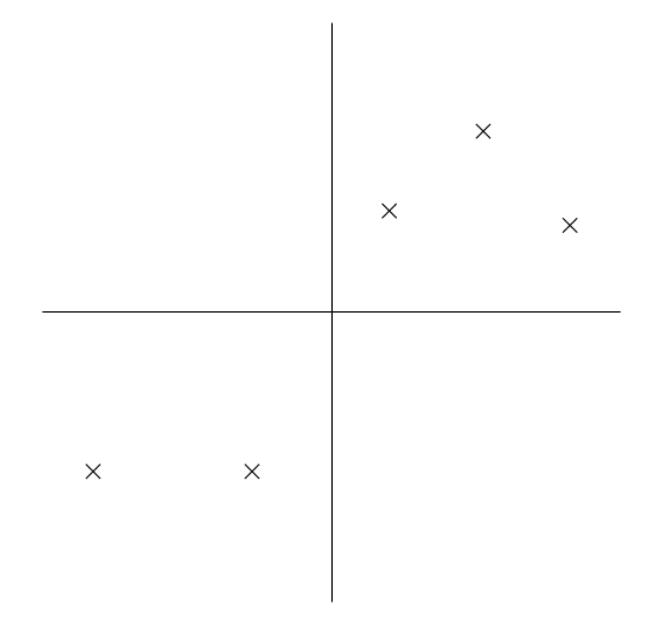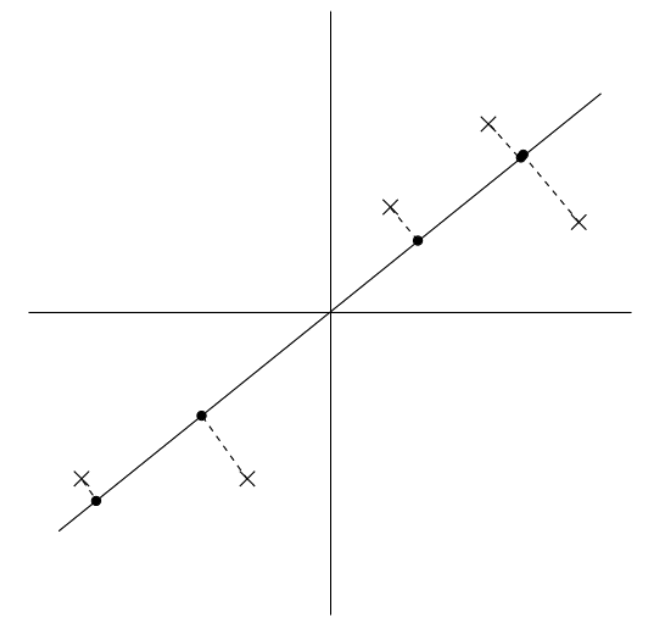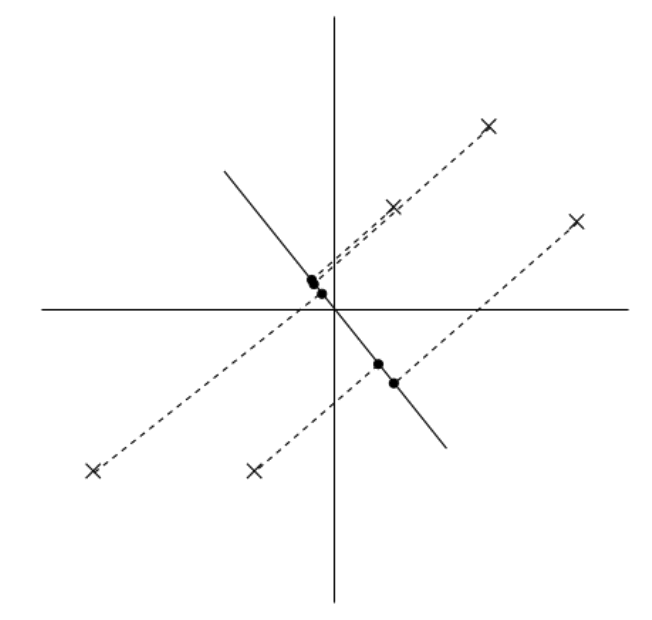
Se pueden visualizar transformaciones o la reconstrucción aproximada:
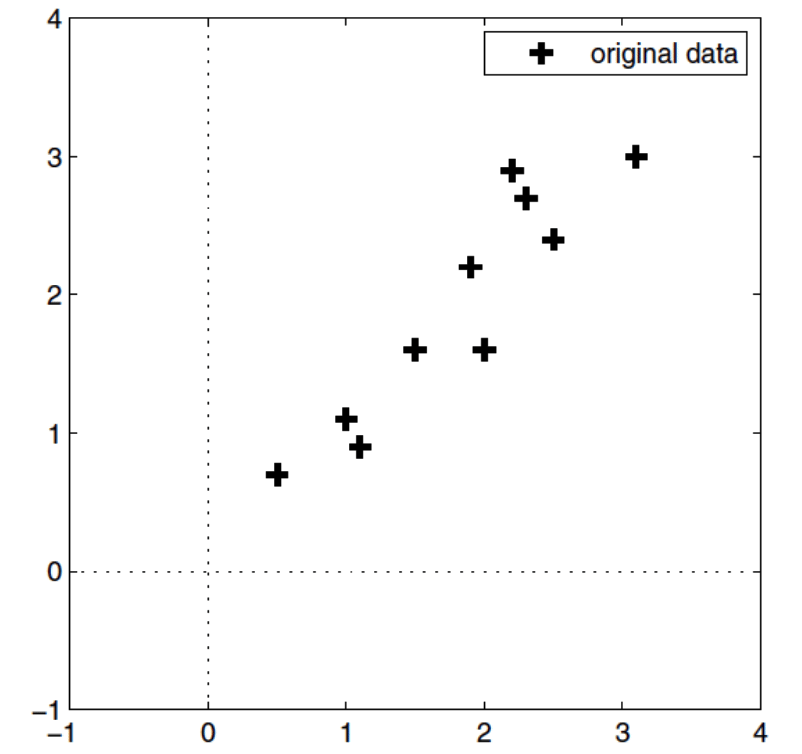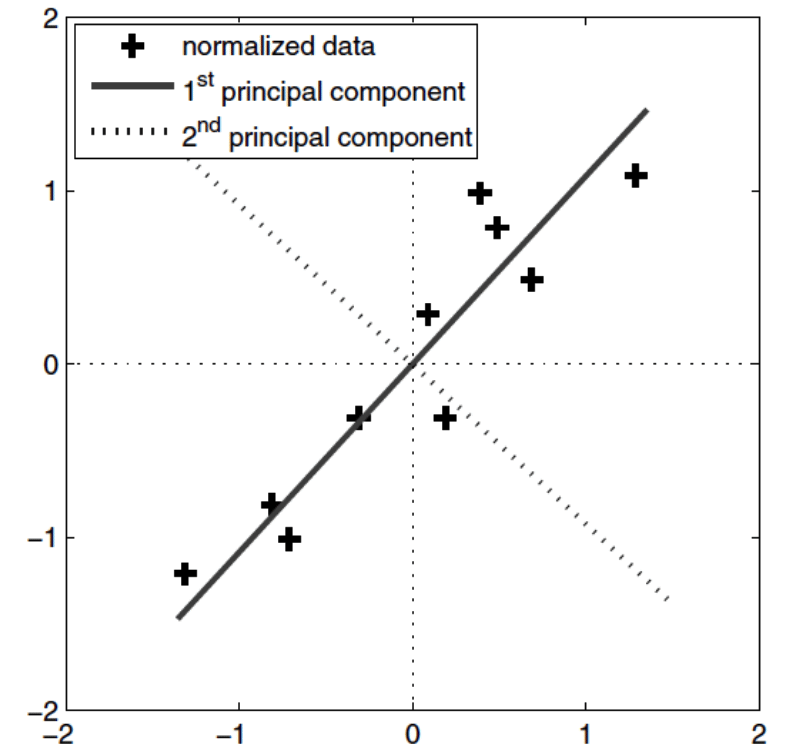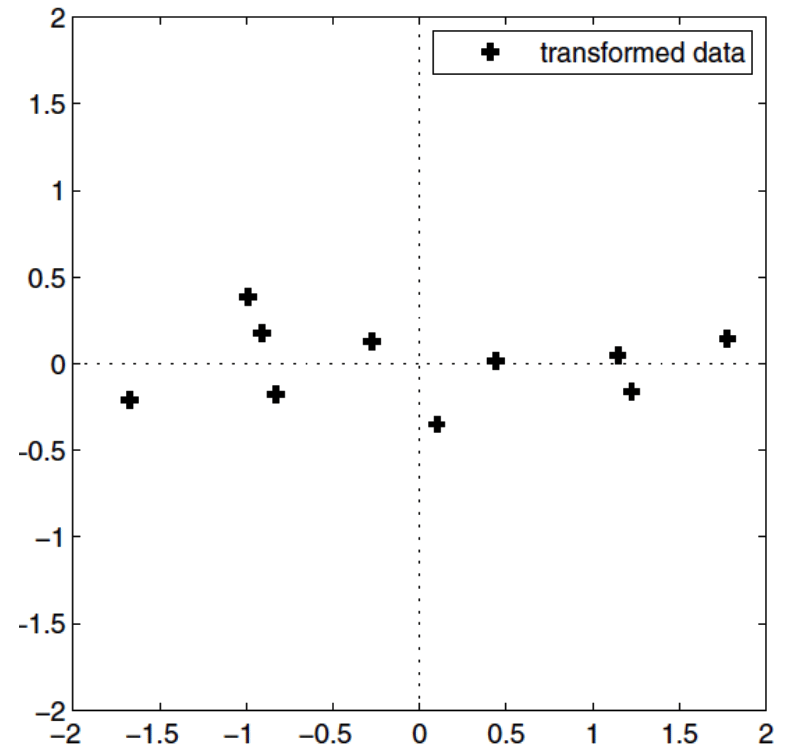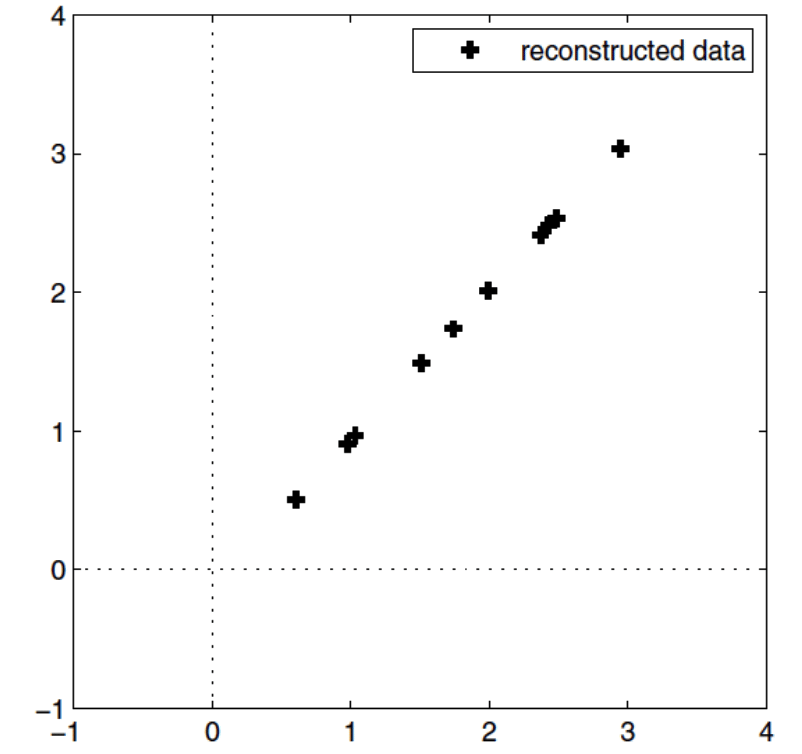
Fundamento Matemático
- Normalizamos \(\mathbf{X}\) a \(\mathbf{X}_0\) (restando la media, opcionalmente dividiendo por la desviación estándar).
- Calculamos \(\Sigma = \mathbf{X}_0^\top \mathbf{X}_0\) (matriz de covarianza).
- Hallamos vectores propios \(u_k\) y valores propios \(\lambda_k\) de \(\Sigma\).
- Elegimos los \(L\) vectores con mayores \(\lambda_k\) para capturar, p. ej., 99% de la varianza.
- Proyectamos los datos en esos vectores: se obtiene la representación de \(\mathbf{X}_0\) en dimensión \(L\).
PCA pasos:
1) Centrar datos
2) Calcular covarianza
3) Eigen-decomposition
4) Elegir top L componentes
5) Proyectar / Reconstruir
Ejemplos Visuales
Ejemplo con 32 imágenes y Reconstrucción con 4 componentes

Reconstrucción de la imagen desde nuestros 4 vectores base \(\mathbf{u}_i, i=1...4\). La combinación lineal se calculó de la siguiente manera:
\[ 0,078 \mathbf{u}_1 + 0,062 \mathbf{u}_2 - 0,182 \mathbf{u}_3 + 0,179\mathbf{u}_4 \]
En imágenes de caras (eigenfaces):

Vectores Propios y Covarianza
Para reducir la dimensión con PCA, se calcula la \textbf{matriz de covarianza} de los datos centrados y se obtienen sus \textbf{valores y vectores propios}. Recordemos:
\[ \text{cov}(X,Y) \;=\; \mathbb{E}\bigl[(X - \mathbb{E}[X])(Y - \mathbb{E}[Y])\bigr]. \]- \(\text{cov}(X,Y) > 0\) indica relación lineal directa (si \(X\) aumenta, \(Y\) también).
- \(\text{cov}(X,Y) < 0\) indica relación lineal inversa.
- \(\text{cov}(X,Y) = 0\) sugiere ausencia de relación lineal aparente.
En \textbf{matriz de covarianza} \(\mathbf{\Sigma}\) (dimensión \(d\times d\)), cada entrada \(\Sigma_{ij}\) es la covarianza entre el atributo \(i\) y el atributo \(j\). Si tenemos datos centrados \(\mathbf{X}_0\), se define:
\[ \mathbf{\Sigma} \;=\; \mathbf{X}_0^\top \,\mathbf{X}_0 \quad (\text{o bien } \tfrac{1}{n}\mathbf{X}_0^\top \mathbf{X}_0 \,\text{dependiendo de la convención de normalización}). \]Los \textbf{valores propios} \(\lambda\) y \textbf{vectores propios} \(\mathbf{u}\) de \(\mathbf{\Sigma}\) satisfacen
\[ \mathbf{\Sigma}\,\mathbf{u} \;=\; \lambda \,\mathbf{u}. \]Los \(\lambda\) más grandes corresponderán a las \textbf{direcciones de mayor varianza} en los datos.
PCA: Aproximación de los Datos
En PCA, se busca aproximar el conjunto de datos \(\mathbf{X}\) con un subespacio de dimensión \(L\). Sean \(\{\mathbf{b}_i\}_{i=1..L}\) vectores ortonormales que generan ese subespacio (los ejes principales). El error de proyección se minimiza cuando:
\[ (\mathbf{b}_i)_{i=1..L} \;=\; \underset{(\mathbf{b}_i)_{i=1..L}}{\arg\max}\; \sum_{i=1}^{L}\, \mathbf{b}_i^\top \,\mathrm{cov}(\mathbf{X})\,\mathbf{b}_i. \]- \(\mathrm{cov}(\mathbf{X})\) denota la matriz de covarianza de los datos centrados.
- La mejor base \(\{\mathbf{b}_1, \dots, \mathbf{b}_L\}\) corresponde, en la práctica, a los \(\mathbf{u}_1,\dots,\mathbf{u}_L\) vectores propios de \(\mathrm{cov}(\mathbf{X})\) asociados a los \(\lambda\) más grandes.
En notación concreta:
\[ \mathrm{cov}(\mathbf{X}) \;=\; (\mathbf{X} - \mu_\mathbf{X})^\top (\mathbf{X} - \mu_\mathbf{X}), \]donde \(\mu_\mathbf{X}\) es la media de \(\mathbf{X}\).
PCA: Normalización (Centrado) de los Datos
Sea \(\mathbf{X}\) una matriz \(d \times n\) (d: número de atributos, n: número de ejemplos). Para construir \(\mathbf{X}_0\) centrada:
- Calcule la media de cada columna (atributo): \[ \mu_i = \tfrac{1}{d}\,\sum_{k=1}^d \mathbf{X}_i^{(k)} \quad\bigl(\text{o a veces} \tfrac{1}{n}\,\sum_{k=1}^n \text{depende de filas/columnas}\bigr). \]
- Reste esa media a la columna: \[ \mathbf{X}_0 = \mathbf{X} - \mu. \]
En cada columna, la suma (o media) pasa a ser 0, eliminando el \textbf{sesgo}. Opcionalmente, se divide además por la desviación estándar para escalarlos. Así, evitamos que atributos con escalas grandes dominen la varianza total.
Xb -> (Xb - media_col)
-> (opcional) / desviacion_col
PCA: Principio y Método
Dado un dataset \(\mathbf{X}\), el algoritmo PCA se realiza típicamente así:
Crear la matriz de datos \(\mathbf{X}\) (dimensión \(d \times n\)), donde cada columna representa un ejemplo (o cada fila, según convención).
Centrar los datos en \(\mathbf{X}_0\):
\[ \mathbf{X}_0 = \mathbf{X} - \mu \]\(\mu\) es la media por atributo (o por componente), de modo que la media de cada columna quede en 0.
(Opcionalmente, se puede escalar además por la desviación estándar para igualar las escalas.)Calcular la matriz de covarianza:
\[ \mathbf{\Sigma} = \mathbf{X}_0^\top \,\mathbf{X}_0 \](o a veces dividido por \(n\), según la convención estadística).
Obtener los vectores propios \(\mathbf{u}_k\) y valores propios \(\lambda_k\) de \(\mathbf{\Sigma}\):
\[ \mathbf{\Sigma} \mathbf{u}_k = \lambda_k \mathbf{u}_k. \]Ordenar los vectores propios en orden decreciente de \(\lambda_k\).
Elegir los \(L\) vectores con valores propios más grandes, cumpliendo por ejemplo que
\[ \frac{\sum_{k=1}^{L}\,\lambda_k}{\sum_{k=1}^{n}\,\lambda_k} > 0.95 \quad (\text{o el umbral deseado de varianza explicada}). \]Proyectar los datos sobre esos \(L\) vectores para obtener una representación de dimensión menor (\(\mathbb{R}^L\)). Cada ejemplo se convierte en \(\mathbf{u}_1^\top \mathbf{X}_0, \dots, \mathbf{u}_L^\top \mathbf{X}_0\).
(Opcional) Reconstruir los datos en la dimensión original si se desea una aproximación de \(\mathbf{X}\), sabiendo que hay una pérdida de información si \(L < d\).
Intuición: Máxima Varianza
La idea fundamental de PCA es buscar direcciones (vectores) que maximicen la varianza de la proyección. En particular, la primera componente principal es:
\[ \max_{\|\mathbf{u}\|=1} \ \mathbf{u}^\top \bigl(\mathbf{X}_0^\top\,\mathbf{X}_0\bigr)\, \mathbf{u}, \]y la \(\mathbf{u}\) que soluciona esto es el vector propio de \(\mathbf{\Sigma} = \mathbf{X}_0^\top \mathbf{X}_0\) con mayor valor propio \(\lambda\). Después, la segunda componente es la dirección ortogonal a la primera que maximiza la varianza restante, y así sucesivamente.
La proyección en los primeros \(L\) vectores propios captura buena parte de la varianza total, reduciendo la dimensión mientras preserva la mayor información posible.
Otros Algoritmos
- Multidimensional Scaling (MDS): Preserva distancias entre puntos en la proyección.
- t-SNE: Modela la cercanía de puntos en alta dimensión a una proyección 2D:
- ICA (Independent Component Analysis): Busca componentes estadísticamente independientes.
- UMAP: Se basa en geometría riemanniana y topología algebraica:
- Autoencoder: Red neuronal no supervisada para comprimir (encoder) y reconstruir (decoder), usando la capa latente como reducción de dimensión.
Conclusiones
- Selección de atributos (filtrada o basada en un modelo) reduce complejidad y puede mejorar rendimiento, sobre todo en métodos sensibles a ruido y correlaciones.
- Reducción de dimensionalidad (PCA, t-SNE, UMAP, etc.) proyecta los datos a un espacio de menor dimensión, lo que facilita la visualización, disminuye ruido y puede acelerar el entrenamiento.
- Hay que equilibrar la simplicidad lograda y la pérdida de información al desechar o proyectar atributos.